Krill, il piccolo gamberetto antartico dal grande genoma

Il krill antartico (Euphausia superba) è l’organismo animale più abbondante sul pianeta, con una biomassa totale compresa tra i 300 e i 500 milioni di tonnellate. Questo piccolo gamberetto riveste un ruolo vitale per l’ecosistema antartico poiché rappresenta il principale collegamento tra i produttori primari che compongono il fitoplancton e i livelli più alti della catena alimentare come uccelli marini, foche, pinguini e balene. Grazie alla sua enorme biomassa, il krill incide in modo significativo su fondamentali processi biogeochimici globali quali i cicli del carbonio e il riciclo del ferro: studiarne la biologia e comprenderne le potenzialità di adattamento a un ambiente in continua evoluzione a causa degli effetti del riscaldamento globale risulta, quindi, fondamentale. Fino ad ora, l’impossibilità di ricostruire la sequenza del genoma di krill – ben 15 volte più grande di quello umano – ha rappresentato un ostacolo tecnico insormontabile per l’approfondimento degli aspetti fisiologici, molecolari e genetici del krill.
Nella ricerca The enormous repetitive Antarctic krill genome reveals environmental adaptations and population insights, pubblicata su Cell e firmata da Changwei Shao del Yellow Sea Fisheries Research Institute di Qingdao in collaborazione con un team di ricercatori del dipartimento di Biologia dell’Università di Padova – composto da Cristiano De Pittà, Gabriele Sales e Alberto Biscontin (ora all’Università di Udine) –, grazie alle più innovative tecnologie di sequenziamento e ai più aggiornati metodi di analisi è stato possibile ricostruire per la prima volta l’intero genoma di krill che risulta essere il più grande di tutto il mondo animale mai sequenziato fino ad ora. Le sue enormi dimensioni sembrano essere il risultato della duplicazione e spostamento di numerosi segmenti di DNA – gli elementi trasponibili – in posizioni diverse del genoma, e non di duplicazioni dell’intero genoma, come osservato in altre specie.
Abbiamo approfondito lo studio con Cristiano De Pittà, docente del dipartimento di Biologia dell’Università di Padova e co-autore della ricerca.
Professore, come si è avvicinato al krill? Cosa l’ha portata a studiare questo minuscolo gamberetto antartico?
Mi sono avvicinato allo studio del krill ancora nel 2006-2007, quindi un bel po’ di tempo fa, perché, sebbene venisse molto studiato da un punto di vista ecologico e fisiologico, da un punto di vista trascrittomico, cioè di espressione dei geni – quali e quanto sono accesi o sono spenti determinati geni –, non si sapeva assolutamente nulla. Il primo lavoro è stato quello di clonare le sequenze dei geni: in gergo si dice “fare delle librerie”. È come se io creassi una libreria in cui i libri corrispondono alle sequenze dei geni che vengono espressi e poi tradotti in proteine, quindi l’informazione più importante che viene utilizzata dalla cellula.
Dopo questo primo lavoro in cui abbiamo cominciato a definire il trascrittoma del krill, abbiamo avviato un’importante collaborazione con la professoressa Meyer dell’Università di Oldenburg e dell’AWI Institute di Bremerhaven, l’istituto polare tedesco più importante che possiede una nave oceanografica molto grande.
Insieme a loro abbiamo partecipato a un progetto di cinque anni, finanziato dall’Helmholtz Institute, che si chiamava Polar Time e che aveva come scopo quello di capire come variasse il ritmo circadiano in questi organismi che sono esposti a regimi di luce costante e fotoperiodi estremi. Grazie a questa collaborazione siamo andati avanti nel clonaggio di questi geni e abbiamo definito per la prima volta il trascrittoma diurno del krill, in condizioni naturali, fornendo una prima caratterizzazione della regolazione genetica dei cambiamenti fisiologici che si verificano durante le 24 ore di una giornata estiva antartica. Abbiamo osservato che durante le diverse ore di luce alcuni geni presentavano dei profili di espressione ciclici o sinusoidali.
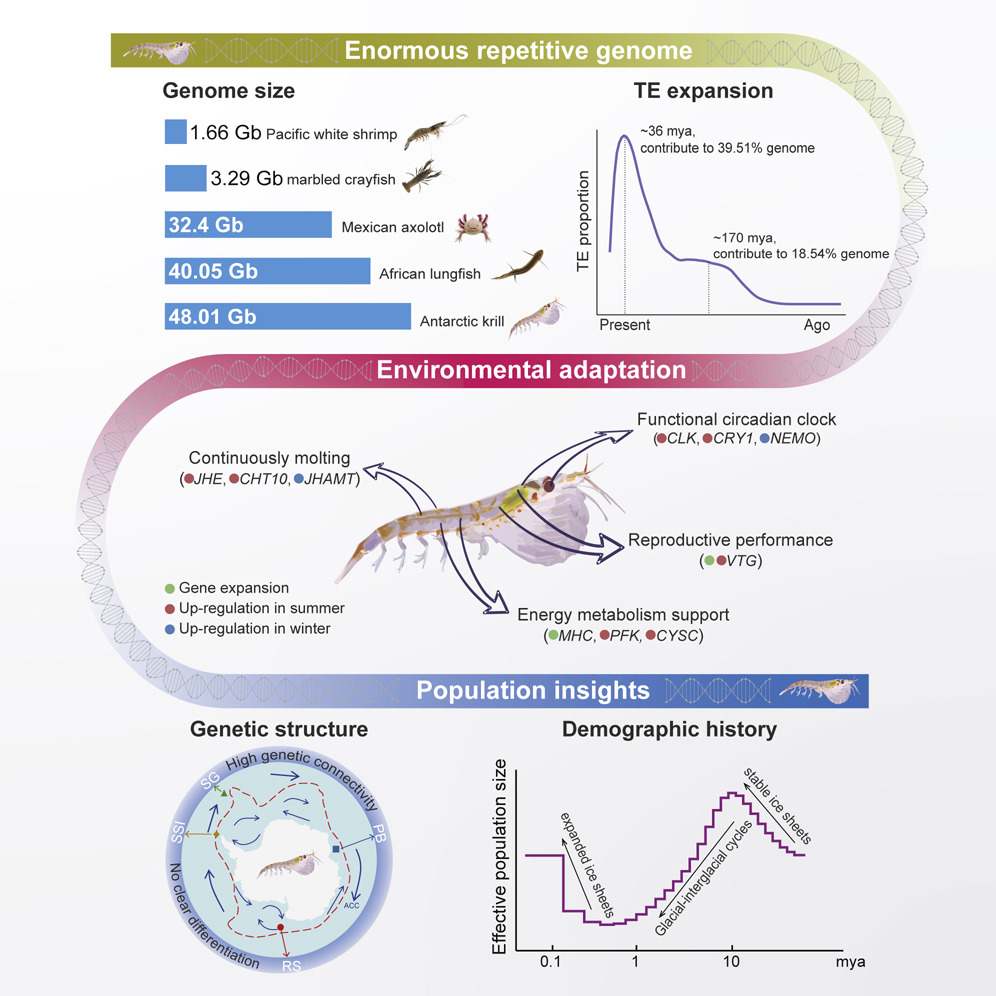
Il passaggio successivo è stato quello di vedere come variavano questi geni in una condizione di laboratorio; per farlo, abbiamo collaborato con il professor Kawaguchi del Department of Environment and Heritage dell’Australian Antarctic Division, il direttore dell’unico istituto al mondo in cui è possibile allevare i krill, in Tasmania. Abbiamo eseguito degli esperimenti nelle 24 ore in condizioni controllate sia di luce e buio alternati che di buio costante: questo ci ha permesso di osservare che effettivamente c’è un orologio biologico funzionante all’interno del krill che è in grado di controllare e orchestrare l’espressione di centinaia di geni. Tutti i dati di trascrittomica sono stati raccolti in un database, il KrillDB2,sviluppato dal professor Gabriele Sales – che è un bioinformatico – e dai suoi collaboratori.
Successivamente, abbiamo identificato da un punto di vista molecolare, con il contributo fondamentale del dott. Alberto Biscontin, quali sono gli attori principali di questo orologio biologico che sono ben noti e ben caratterizzati, ad esempio, nella drosofila – il moscerino della frutta –, nel topo e nell’uomo.
Abbiamo clonato i geni-orologio di krill e adesso stiamo procedendo con l’identificazione e clonaggio di altri geni che regolano il ritmo circadiano. Nel frattempo siamo venuti in contatto con il professor Shao di un consorzio cinese che aveva cominciato a sequenziare il genoma che – come si è visto dalla pubblicazione – era stimato essere enorme, quindi lo sforzo è stato veramente titanico da parte loro in termini di sequenziamento e assemblaggio delle sequenze.
Grazie alla collaborazione con lui e all’apporto del professor Sales, del dottor Biscontin e mio siamo riusciti nell’interpretazione biologica dei dati, in particolare legati alla parte circadiana: abbiamo capito, infatti, che molti dei geni che trovavamo essere ritmici nel gamberetto, sia in condizioni naturali che di laboratorio, presentano delle sequenze di riconoscimento per i geni-orologio (chiamati “clock”). È stata un’ulteriore conferma del fatto che l’orologio endogeno esiste ed è funzionante.
Ora che la sequenza è nota, sarà possibile approfondire con studi molecolari il funzionamento di determinati geni anche sulla base delle condizioni ambientali che cambiano, quindi si riuscirà a capire meglio quali sono e come vengono controllati i loro livelli di espressione.
Sembra incredibile che esseri così piccoli nascondano un genoma così grande. Ve lo aspettavate?
È stato incredibile anche per noi! Quando sono uscite le prime stime, mi sembrava quasi impossibile. Sapevamo solo che il genoma era molto grande, ma effettivamente non sapevamo da che cosa fosse dovuta questa enorme dimensione: senza avere la sequenza era impossibile capire a che cosa fosse dovuto. Quando abbiamo scoperto che il genoma era effettivamente 15 volte più grande di quello umano, abbiamo subito pensato che ci fosse anche una duplicazione dell’intero genoma.
Mi spiego meglio: anche nelle piante si parla spesso di poliploidia, cioè del fatto che tutto il genoma è suddiviso in cromosomi, i quali vengono duplicati. È chiaro che questo raddoppia la quantità di DNA genomico: nel krill, in realtà, non è proprio così. Ci sono questi segmenti di DNA, noti come trasposoni, che vengono duplicati – quindi sì, la stessa sequenza si duplica e diventa doppia – ma la sequenza doppia si trasferisce in un altro punto del genoma: tutte queste ripetizioni hanno portato il genoma a ingrandirsi tantissimo. Nello studio hanno ipotizzato che questi eventi di duplicazione siano stati legati a due grandi eventi di cambiamento climatico e si siano innescati in seguito a una situazione di stress.
Quanto è importante in questo ambito la ricerca internazionale, la collaborazione con altri istituti e università?
È fondamentale. Lo sforzo in termini di sequenze prodotte è stato enorme e lo sforzo maggiore, anche in termini economici, lo ha fatto il gruppo cinese anche perché aveva accesso a dei centri di sequenziamento – come il Bejing Institute, a Pechino – con un grande numero di sequenziatori di nuova generazione che permettono di produrre miliardi di sequenze contemporaneamente. Questi strumenti permettono di effettuare il sequenziamento massivo parallelo o Next Generation sequencing, ossia tecniche che dal 2004 in poi hanno sostituito i vecchi metodi e permettono di produrre una mole di dati impressionante con costi molto più bassi rispetto al passato.
Il problema di questa mole di dati è che poi è necessario che tutte queste sequenze vengano assemblate, ossia si incastrino l’una con l’altra, proprio come i tasselli di un puzzle, per dare l’immagine finale del genoma. Lo sforzo per mettere in ordine tutti questi tasselli e far risultare una sequenza unica è stato davvero enorme, soprattutto perché esistono delle sequenze che combaciano con più di una: è come se un pezzo del puzzle potesse incastrarsi con più tessere diverse. Il problema delle sequenze ripetute è ovviamente la difficoltà più grande in un processo di assemblaggio genomico perché bisogna cercare di capire qual è il “partner” esatto di queste tessere così simili tra di loro.
Sono state utilizzate due tecniche di sequenziamento: una che permette di ottenere sequenze della lunghezza di 150 basi (sequenze piuttosto corte), e un’altra che si chiama Pacific Bioscience che permette di ottenere sequenze lunghe anche migliaia di basi; combinando i due approcci è stato possibile riscostruire il puzzle anche con le tessere più “ambigue”.
Mi sembra di capire che uomo e macchina siano complementari: il ruolo dei sequenziatori è indispensabile, ma anche la mente che interpreta i dati.
Certo. Ci vogliono degli algoritmi, strumenti informatici molto avanzati, e poi bisogna risolvere le situazioni ambigue una per una. Il problema attuale non è tanto quello di produrre sequenze di DNA, perché in questo momento è facile e ne possiamo produrre tantissime anche a basso costo. Il problema è mettere insieme tutte queste informazioni e dare un senso a quello che produciamo.
In merito alle prospettive future della ricerca sul lato pratico, si può dire che gli esseri umani dovrebbero “imparare” dal krill su come adattarsi a queste estreme variazioni stagionali?
Come è successo per il genoma umano, sapere quali sono i geni presenti in un genoma è solo il punto di partenza per capire come questa informazione genetica verrà utilizzata dall’organismo per adattarsi a condizioni ambientali che cambiano nel tempo – dovute per esempio al surriscaldamento, ai cambiamenti della composizione delle acque, eccetera –, e quindi ci permetterà di capire meglio quali meccanismi biologici si attivano in determinate condizioni. Grazie alla genomica funzionale sarà possibile studiare sia i normali processi biologici che permettono la sopravvivenza del krill in un ambiente così estremo sia il suo adattamento a condizioni di stress.









