Scienza e informatica: dieci codici che hanno aiutato la ricerca

“In qualsiasi ramo della ricerca scientifica, dietro c’è un computer”: così riporta un recente articolo pubblicato su Nature in cui vengono elencati i 10 codici che hanno aiutato la ricerca e la scienza negli anni passati. Oggi, infatti, si utilizzano numerosi codici per implementare, velocizzare, confrontare, elaborare e codificare dati, calcoli, testi, immagini e tutto ciò che è necessario all’indagine scientifica. Dai database alla programmazione di modelli fino alle operazioni di calcolo e ai programmi di visualizzazione e analisi delle immagini: questo insieme di algoritmi e software ha permesso di creare il legame ormai essenziale tra computer science e ricerca scientifica, fondamentale oggi in tutti i campi. Ma, come scrive l’autore dell’articolo, dietro a questo progresso c’è sempre bisogno del pensiero “umano”.
Gli anni Cinquanta e Sessanta, dalla meteorologia al DNA
Il viaggio temporale proposto si può leggere su due livelli: il primo riguarda il boom informatico a partire dal secondo dopoguerra a oggi; mentre il secondo livello di lettura ci aiuta a dare uno sguardo diverso alla scienza, mettendo in luce quali sono stati gli ambiti scientifici che sono stati al centro della ricerca nei vari decenni (e che quindi hanno utilizzato la computer science per implementare le diverse attività).
Dagli anni Sessanta a oggi, la memoria dei computer è aumentata di ben 10.000 volte. Questo non ha fermato, tuttavia, i programmatori e i ricercatori dell’epoca nello sviluppo di codici per raggiungere gli obiettivi. Uno dei primi linguaggi di programmazione è il Fortran (FORmula TRANslation), apparso nel 1957: il lavoro è stato svolto da un team della IBM, guidato da John Backus ,che era alla ricerca di un linguaggio semplice, più vicino alla semantica del linguaggio naturale. Veniva utilizzato un compilatore, un macchinario che traduceva il linguaggio di programmazione in un altro (in questo caso da formule matematiche a codici), attraverso l’uso di schede perforate a 80 colonne.

Una scheda perforata Fortran
L’obiettivo era velocizzare i programmi che usavano molti algoritmi e calcoli matematici: codificandoli, tuttavia, i tempi diminuirono e la programmazione diventò parte integrante del lavoro, in particolare nella creazione di modelli climatici, nella dinamica dei fluidi e nella chimica computazionale. Sempre di traduzione si tratta anche la Trasformata veloce di Fourier, un algoritmo utilizzato per ottimizzare e ridurre il numero di operazioni richieste: venne applicata per accelerare i calcoli utilizzati per determinare la natura delle onde radio, trasformandole in funzioni di frequenza. Ancora più interessante, soprattutto perché diventò la base della bioinformatica, è la creazione di una serie di database interconnessi e organizzati in modo razionale in cui vennero inseriti i dati relativi alla struttura di 65 proteine. Questo fu realizzato da Margaret Dayhoff nel 1965, con l’obiettivo di confrontare le varie sequenze di amminoacidi alla ricerca delle relazioni evolutive tra le specie. Per questo sviluppo un database molecolare accessibile dal. computer o tramite la linea telefonica che si collegava a dei computer in remoto.
Meet Margaret Dayhoff: Bioinformatics pioneer & BLOSUM matrix creator #DailyScienceSpark https://t.co/W3DRlV2nIu pic.twitter.com/Fb5FjdyFmQ
— Paperpile (@paperpile) May 30, 2016
Velocità, open source e immagini: nuovi percorsi per la computer science
Fino ad ora, le novità della computer science applicata alla ricerca si basavano sull’utilizzo di computer, strumenti e macchinari già esistenti, adattando le loro funzionalità alle necessità degli scienziati. Verso la fine degli anni Sessanta, tuttavia, il matematico John Von Neumann riadatta i calcolatori utilizzati durante la Seconda guerra mondiale per tracciare le traiettorie balistiche in strumenti per le previsioni climatiche, sfruttando le leggi della fisica. Per riuscirci, data la quantità enorme di calcoli, era necessario modificare i computer: per velocizzare il processo, von Neumann mise i dati e le istruzioni del programma nella stessa memoria. BLAS, Basic Linear Algebra Subprograms, fu un interfaccia utilizzata per le operazione ad alta intensità di calcolo di matrici e vettori: la novità, in questo caso, è la capacità di essere utilizzata su qualsiasi computer. Questo ha dato la possibilità anche ai produttori di hardware di implementare le loro macchine affinché BLAS lavori più rapidamente.
Sempre di open source si tratta anche il programma di elaborazione e analisi delle immagini NIH Image, oggi ImageJ, pioniere di tutti i software legati al mondo della fotografia. l’autore è Wayne Rasband, programmatore all’epoca (siamo nel 1987) del National Institute of Health degli Stati Uniti. Il software gratuito, creato su uno dei primi Macintosh della Apple, prevedeva la possibilità di compiere una serie di operazione, dalla calibrazione spaziale all’analisi automatica delle particelle, su immagini di natura scientifica, avendo anche la capacità di essere compatibile con altri programmi, come quelli per scannerizzare. L’interfaccia era personalizzabile e si potevano aggiungere diversi plug-in a seconda delle esigenze.
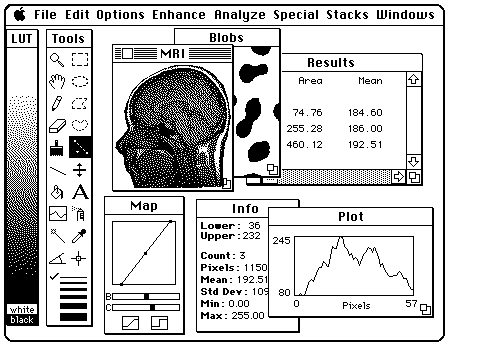
Dal manuale di NIH Image
Gli anni Novanta e l’inizio dei Duemila: è tempo di condivisione
Uno dei punti fondamentali della ricerca scientifica è la condivisione delle informazioni e con l’avvento di internet questo passaggio è diventato essenziale per il progresso e lo sviluppo di tutte le scienze. Un esempio è BLAST, un algoritmo poi diventato programma che il New York Times ha definito nel 1990 “il Google della ricerca biologia”. Sostanzialmente la funzione principale di BLAST era il confronto delle informazione delle sequenze genetiche, presenti nei database. La velocità di ricerca è data dall’approccio euristico del programma: data la crescente raccolta di dati, la ricerca di una corrispondenza esatta era esageratamente lunga. Il metodo alla base di BLAST è di trovare velocemente una soluzione approssimativa, calcolando anche la casualità delle corrispondenze, dando la possibilità di ampliare lo sguardo sui dati ai ricercatori.
Come detto poco fa, il confronto fra i ricercatori, anche di diverse discipline, è di vitale importanza oggi: uno dei primi pionieri di questo approccio è l’archivio arXiv, fondato nel 1991 e ancora oggi attivo sul web. Si tratta di una raccolta di pre-prints (bozze definitive) di articoli di carattere scientifico ed economico: a oggi, il numero di lavori presenti è di circa 1,8 milioni. Non sono presenti revisioni alla pari, peer review: il sistema offerto è l’endorsment: pur mantenendo un livello minimo di qualità, l’articolo viene approvato attraverso il giudizio di un autore certificato oppure in maniera automatica.
Altro passo in avanti della ricerca scientifica è l’elaborazione dei dati: Notebook IPhyton, nato dalla precedente versione del 2001, è un software gratuito per l’elaborazione interattiva in cui si possono combinare più elementi come codici, testi, calcoli, grafici e media in un unico spazio, grazie anche alla possibilità di modulare lo spazio di lavoro e adattarlo alle proprie esigenze attraverso plug-in. Oggi Notebook Iphyton si è evoluto in Project Jupiter, sviluppato sul web.
Una delle frontiere della scienza più interessanti è sicuramente l’intelligenza artificiale. Alex Net fu nel 2012 la prima rete neurale convoluzionale che utilizzò la GPU per potenziare le prestazioni: queste reti neurali artificiali servono principalmente per il riconoscimento e l’identificazione di immagini e video attraverso delle connessioni su più livelli. Attraverso il database ImageNet, il pre addestramento e l’impiego di hardware per l’elaborazione video su pc, questa architettura di rete neurale profonda 8 livelli è riuscita a velocizzare questi processi, facendo vincere ai Alex Krizhevsky e Ilya Sutskever di vincere la competizione mondiale nel 2012 di computer vision.
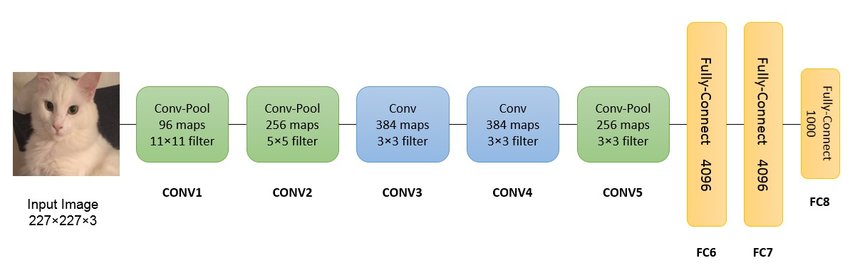
L'architettura di Alex Net semplificata









